LAMMPS - i od razu jaśniej
Dodane przez piotr dnia 19.02.2014 20:09
Była już u nas mowa o systemach obliczeniowych dynamiki cząstek (patrz news o metodach bezsiatkowych ). Teraz trochę o jednym z najlepszych systemów tego typu opartym na licencji MIT. Mowa o LAMMPS - Molecular Dynamics Simulator.
LAMMPS został pierwotnie opracowany w ramach amerykańskiego Departamentu Energii przez CRADA (Cooperative Research and Development Agreement) złożony z dwóch laboratoriów DOE i 3 firm. Jest on rozprowadzany przez Sandia National Labs .
W najbardziej ogólnym sensie, LAMMPS integruje równania ruchu Newtona dla zbiorów atomów, cząsteczek lub makroskopowych cząstek, które oddziałują za pośrednictwem sił krótkiego lub dalekiego zasięgu z wieloma różnymi warunkami początkowo-brzegowymi.
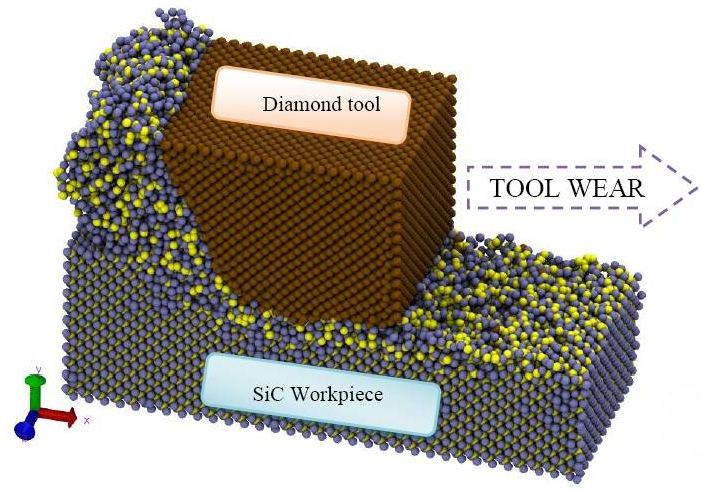
Obszary zastosowań są doprawdy bardzo szerokie (polecam stronę Sandia labs z przykładami).
Rozszerzona zawartość newsa
LAMMPS został pierwotnie opracowany w ramach amerykańskiego Departamentu Energii
W najbardziej ogólnym sensie, LAMMPS integruje równania ruchu Newtona dla zbiorów atomów, cząsteczek lub makroskopowych cząstek, które oddziałują za pośrednictwem sił krótkiego lub dalekiego zasięgu z wieloma różnymi warunkami początkowo-brzegowymi. Dla podniesienia wydajności obliczeniowej LAMMPS tworzy tzw. "listy sąsiadów", dzięki którym śledzić może układ wzajemny cząstek. Listy te służą zoptymalizowaniu gęstości cząstek na małych dystansach, tak że lokalna gęstość cząstek nie jest nigdy zbyt duża. W maszynach równoległych LAMMPS wykorzystuje techniki przestrzennego rozkładu cząstek, aby symulacje podzielić na mniejsze domeny 3D, z których każda jest przypisana do innego procesora. Procesory komunikują się i przekazuj sobie jedynie informacje o cząstkach sąsiednich subdomen leżących na granicy domeny. LAMMPS jest najbardziej skuteczny (w sensie obliczeń równoległych) dla systemów, których cząstki wypełniają prostokątne pudełko z grubsza o jednolitej gęstości.
Wszystkich zainteresowanych odsyłam do bardzo obszernych materiałów na stronie:
Dodam jeszcze od siebie co ważne w systemach otwartych system posiada pre i post procesor, a właściwie pre i post procesory, gdyż oprócz oprogramowania przypisanego bezpośrednio do pakietu dystrybucyjnego można sobie jeszcze ściągnąć wspomaganie, napisane w pytonie pod bardzo apetyczną nazwą Pizza.py. (http://pizza.sandia.gov/)